Publication trimestrielle du Laboratoire
d'analyse et d'architecture des systèmes du CNRS
© LAAS-CNRS
Les oncoprotéines de la famille Ras sont mutées dans environ 30% des tumeurs humaines. Elles sont alors le marqueur d’un mauvais pronostic car elles augmentent la prolifération des cellules tumorales en restant constamment activées. Malgré leur fort potentiel thérapeutique, elles demeurent «undruggable» à ce jour et trouver un point d’attaque pour les cibler permettrait enfin d’imaginer une nouvelle lignée d’inhibiteurs.
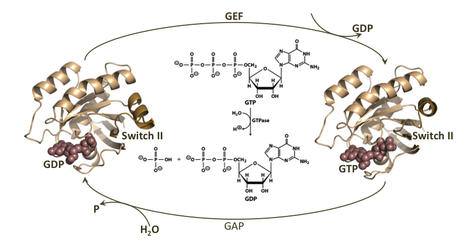
Les oncoprotéines Ras sont des « interrupteurs moléculaires » qui régulent certaines fonctions fondamentales au cœur de la cellule, dont la transduction des signaux de facteurs de croissance. En réponse à des signaux extracellulaires, elles enchaînent des états «off» et «on», en alternant respectivement des phases d’inactivation/activation faisant intervenir l’hydrolyse du GTP en GDP, puis l’échange de GDP/GTP. Lors de ce cycle dont la cinétique est très lente, deux protéines interviennent pour catalyser la réaction et la rendre performante : GAP d’une part active l’hydrolyse, tandis que GEF favorise le relargage du GDP produit, permettant ainsi à une autre molécule de GTP de s’amarrer à Ras pour débuter un nouveau cycle. On sait aujourd’hui que des mutations de cette protéine sont mises en cause dans environ 30% des cancers.
Nous initions, en collaboration avec l’institut Claudius Régaud, une activité d’oncologie structurale mettant en œuvre le calcul au service de cette problématique.
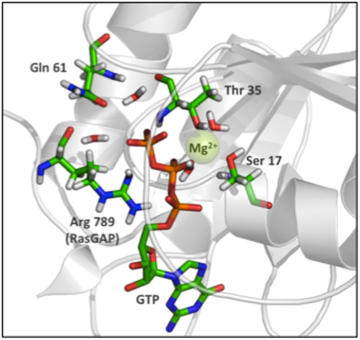
Une fois mutée, Ras n’hydrolyse plus le GTP auquel elle reste lié, bloquée sur le mode « on » induisant à une prolifération anormale des cellules tumorales. Malgré les efforts consentis depuis des dizaines d’années, cette cible thérapeutique majeure demeure un verrou pour la recherche oncologique. La simulation à l’échelle atomique se place alors comme un outil de prédilection pour décrypter les propriétés de Ras avec un précision inégalée, mais aussi pour multiplier les expériences in silico et limiter le coût des expériences in vitro. C’est dans cette optique que nous initions, en collaboration avec l’institut Claudius Régaud, une activité d’oncologie structurale mettant en œuvre le calcul au service de cette problématique.
Décrypter les mécanismes d’action et d’inhibition de Ras
Plusieurs voies peuvent être envisagées pour cibler Ras. Restaurer son activité GTPase, très réduite en présence de mutation, bloquer la formation du complexe Ras-GTP en inhibant l’interaction Ras-GEF, ou bloquer la transduction du signal en inhibant l’interaction de Ras avec ses effecteurs en aval. La première solution s’est jusqu’ici avérée infructueuse car le site actif à cibler est déjà très encombré stériquement.
© Marie-Brut/LAAS-CNRS
Nous nous sommes donc engagés dans les deux autres voies qui demeurent un vrai défi car il s’agit d’inhiber une interaction protéine-protéine à l’aide d’une petite molécule médicament. Notre stratégie est basée sur l’outil FleXible, un logiciel original développé au sein de notre équipe et basé sur la méthode des Modes Statiques. Celle-ci est adaptée aux exigences des biomolécules (flexibilité conformationnelle, interactions avec l’environnement, calcul haut-débit) et permet de caractériser de façon systématique la réponse d’un système d’atomes soumis à des contraintes ou modifications de tout type, internes ou externes. Dans ce cas, il s’agit de caractériser les mécanismes fondamentaux de Ras et de ses mutants : hydrolyse, corrélations internes à distance, interaction entre Ras et ses partenaires amont et aval.
Restaurer l’activité d’ « interrupteur moléculaire » de Ras
Notre projet SAMO (SmArt screening of Mutations for structural Oncology) récemment accepté pour financement dans le cadre de l’appel à projet plateforme du RITC, vise à mettre en œuvre ces simulations pour acquérir une compréhension détaillée des processus biomécaniques qui gouvernent la structure et l’activité de Ras et de ses mutants. En particulier, nous modifions et stimulons Ras in silico pour simuler son mécanisme d’activation/inactivation. Nous cherchons ainsi à cribler et cartographier de possible sites d’attaque qui seront ensuite testés expérimentalement par l’équipe du Pr. Gilles Favre à l’institut Claudius Régaud. A terme, nous espérons pouvoir mettre à disposition de la communauté d’oncologie toulousaine nos outils et notre savoir faire en matière de simulation des biomolécules. Notre objectif est aussi de créer une banque de données contenant l’ensemble des informations ainsi acquises. Combinée à nos outils de simulation, cette biobanque nous positionnera comme plateforme d’oncologie structurale au cœur du paysage toulousain et permettra à l’utilisateur de manipuler virtuellement ces protéines cibles, de créer ses propres expériences virtuelles.





